A la une

Lancées en 2002, les thérapies combinées à base d’artémisinine (ACT) ont révolutionné la prise en charge du paludisme. Or, comme les médicaments qui les ont précédées, les ACT sont désormais confrontées à des problèmes de résistance. Face à l’urgence de trouver de nouveaux antipaludiques, des chercheurs français tentent d’agir sur de nouvelles cibles. Parmi elles, la subtilisine SUB1, une enzyme-clé du cycle de vie parasitaire.
Depuis le début des années 2000, la lutte contre le paludisme a connu d’importants progrès. De 245 millions de cas en 2000, l’Organisation mondiale de la santé (OMS) estimait que ce chiffre est tombé à 230 millions en 2015. Idem pour le nombre de décès, passé de 897.000 en 2000 à 577.000 en 2015. Parmi les raisons de ce succès, un meilleur contrôle des moustiques vecteurs de la maladie, la distribution en masse de moustiquaires imprégnées d’insecticides, mais aussi l’arrivée de traitements très efficaces. Dénommées ACT (pour Artemisinin-based Combination Therapy), ces thérapies reposent sur l’association de dérivés de l’artémisinine et d’autres médicaments antipaludiques.
Isolée au début des années 1970, l’artémisinine est une molécule issue de l’armoise annuelle (Artemisia annua), plante utilisée de longue date par la médecine chinoise. L’arrivée de l’artémisinine dans l’arsenal thérapeutique a donné un nouveau souffle à la lutte contre le paludisme. Car depuis l’isolement de la quinine au début du 19ème siècle, les médicaments développés contre le paludisme, tels que la chloroquine, la sulfadoxine-pyriméthamine et la méfloquine, ont vu leur efficacité rapidement s’amenuiser. En cause, les résistances développées par les agents du paludisme vis-à-vis de ces molécules.
L’efficacité des ACT compromise par les résistances
Or voilà que, depuis quelques années, le même phénomène est à l’œuvre avec les ACT. Que ce soit contre les dérivés d’artémisinine ou contre les médicaments qui leur sont associés, les résistances ont émergé en Asie à la fin des années 2000, et depuis peu en Afrique, compromettant l’efficacité de ces thérapies, et donc la réponse mondiale contre le paludisme. Lors d’une étude publiée en 2016, une équipe britannique a estimé que la généralisation de ces résistances pourrait entraîner jusqu’à 16 millions de nouveaux cas et 800.000 décès additionnels par an en Afrique.
En outre, les progrès obtenus contre le paludisme semblent s’essouffler. Avec 247 millions de cas estimés en 2021, l’épidémie a retrouvé son niveau de l’année 2000, tandis que le nombre de décès est de nouveau à la hausse (619.000 morts en 2021). Plusieurs experts redoutent l’effet du réchauffement climatique, qui accroît l’aire de répartition des moustiques vecteurs. Certes, l’espoir reste de mise, en particulier dans le champ de la prévention : début juillet, l’OMS a annoncé la livraison prochaine d’un premier vaccin contre le paludisme dans 12 pays africains, mais son efficacité apparait limitée.
Reste désormais à trouver des alternatives aux ACT, sans attendre que ces traitements aient atteint un seuil critique de résistance. Pour cela, la communauté scientifique travaille à l’étude de nouvelles cibles thérapeutiques. Parmi les travaux les plus prometteurs, ceux menés sur la subtilisine 1 (SUB1), une enzyme produite par le parasite responsable du paludisme - il existe cinq espèces de Plasmodium infectant l’Homme, dont les principales sont P. falciparum et P. vivax. Décrite en 1998 par une équipe britannique, SUB1 est une enzyme de la famille des protéases à sérine, dont le rôle est d’activer par protéolyse limitée1 d’autres protéines, dont d’autres protéases, impliquées dans la sortie du parasite des cellules humaines infectées.
Empêcher la sortie des mérozoïtes
Avant d’aller plus loin, un bref rappel sur le cycle de vie complexe de Plasmodium : transmis par une piqûre d’anophèle femelle, le parasite, sous forme de ‘sporozoïte’, gagne aussitôt le foie. Une fois entré dans les hépatocytes (cellules constitutives du foie), il y prolifère et s’y différencie en ‘mérozoïtes’. Après avoir provoqué la destruction (lyse de la membrane) des hépatocytes, Plasmodium gagne la circulation sanguine, où il envahit les globules rouges. Il s’y multiplie de nouveau, engendre la lyse de ces cellules pour en sortir, puis pénètre dans d’autres globules rouges (cycle érythrocytaire). Circulant dans le sang, le parasite pourra de nouveau infecter un moustique à l’occasion d’une piqûre. Il s’y reproduira, puis gagnera sous sa forme ‘sporozoïte’ les glandes salivaires de l’insecte, d’où il pourra être transmis à d’autres hôtes humains.
Revenons à SUB1. En clivant différentes protéines, dont d’autres protéases, elle est à l’origine d’une cascade enzymatique2 entraînant la destruction (lyse) des membranes des cellules hôtes infectées, tant les hépatocytes que les globules rouges. D’où l’intérêt de développer des inhibiteurs spécifiques de cette enzyme : en bloquant le site actif de SUB1, ces molécules l’empêcheraient d’agir contre ses substrats naturels. Ce qui, de fait, bloquerait le déclenchement de la cascade enzymatique à l’origine de la lyse cellulaire, empêchant la sortie des parasites hors des hépatocytes et des globules rouges, et interrompant ainsi leur cycle.
C’est à ce travail que se sont attelés des chercheurs français, au sein d’une collaboration regroupant l’Institut des biomolécules Max Mousseron (IBMM, CNRS/université de Montpellier/ENSCM) et l’Unité de microbiologie structurale de l’Institut Pasteur (Paris). La tâche s’avère délicate, tant les prérequis sont nombreux : « cet inhibiteur doit présenter une forte affinité pour l’enzyme, mais il faut aussi qu’il puisse atteindre sa cible in vivo, qu’il traverse les différentes membranes, et qu’il puisse être administré par voie orale », explique Jean-François Hernandez, directeur de recherche à l’IBMM et coordinateur de ce projet, dénommé SPIM, mené en collaboration avec le Dr. Jean-Christophe Barale, responsable du projet à l’Institut Pasteur.
Bloquer spécifiquement le site actif de SUB1
Les chercheurs ont analysé la séquence des divers substrats de l’enzyme, en particulier le site qu’elle reconnaît et qu’elle coupe au sein de ces protéines. « Nous savons où l’enzyme coupe de manière très spécifique. C’est cette attaque-là qu’il s’agit de mimer avec notre inhibiteur. Les composés que nous avons synthétisés et testés sont donc basés sur des substrats naturels. Il s’agit de pseudopeptides de type a-cétoamide, d’une longueur de six acides aminés, au sein duquel la liaison peptidique ciblée par SUB1 est remplacée par une liaison qui réagit avec le site catalytique de l’enzyme mais qui ne peut pas être coupée. Ce sont des inhibiteurs compétitifs, car ils entrent en compétition avec les substrats naturels de SUB1 », explique Jean-François Hernandez.
Les premières étapes de cette longue recherche s’avèrent très concluantes : en août, l’équipe de l’Institut Pasteur a publié la structure 3D de la protéase SUB1 associée à un inhibiteur3.
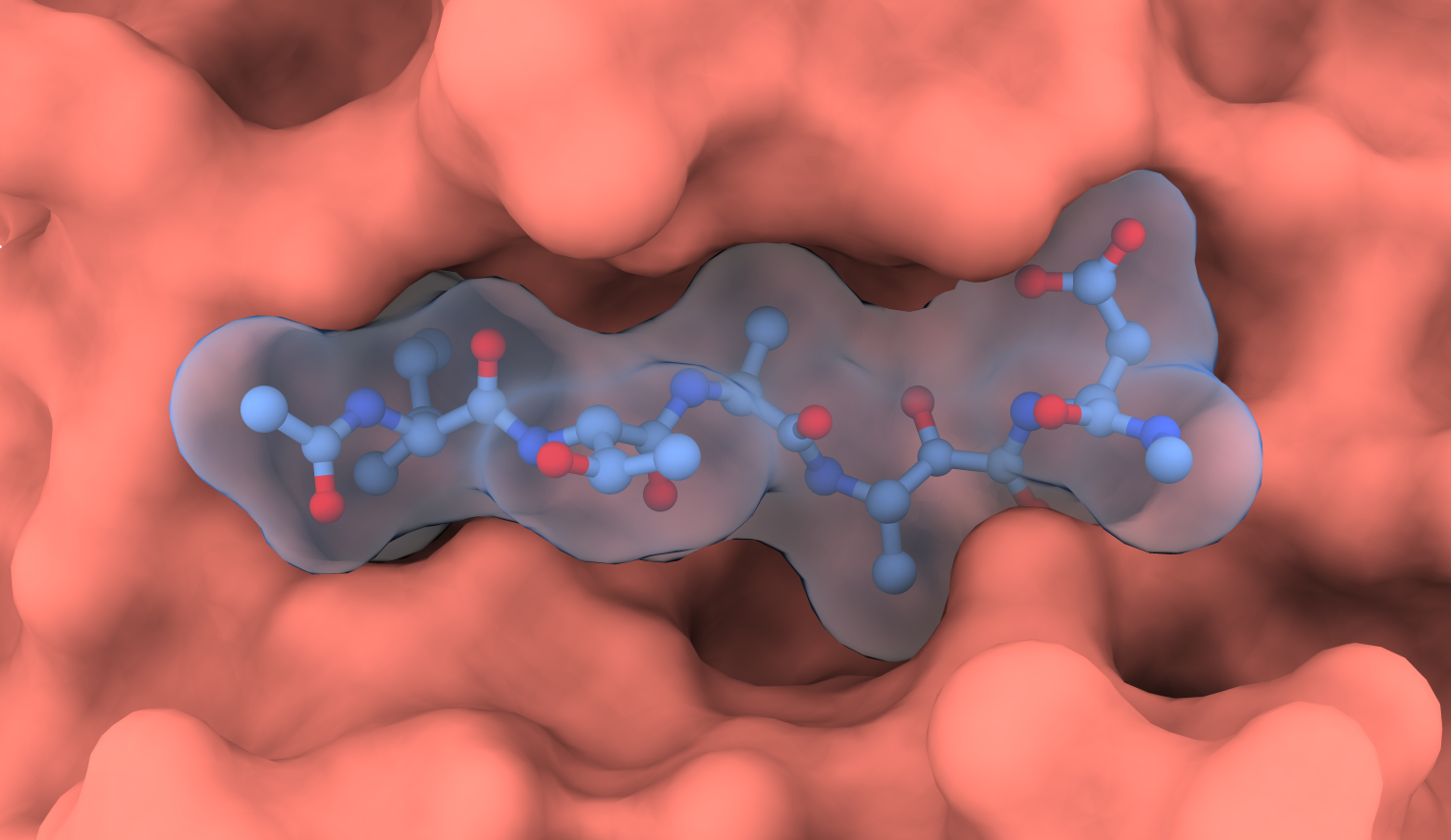 Structure d’un complexe SUB1/inhibiteur : liaison d’un inhibiteur (bâtonnets bleu gris ; boules rouges = oxygène ; boules bleues = azote, avec en transparence la surface atomique) dans le site actif de SUB1 (surface atomique rose) de Plasmodium vivax. La structure tri-dimensionnelle de ce complexe a été résolue par cristallographie aux rayons X.4
Structure d’un complexe SUB1/inhibiteur : liaison d’un inhibiteur (bâtonnets bleu gris ; boules rouges = oxygène ; boules bleues = azote, avec en transparence la surface atomique) dans le site actif de SUB1 (surface atomique rose) de Plasmodium vivax. La structure tri-dimensionnelle de ce complexe a été résolue par cristallographie aux rayons X.4
De plus, les tests d’inhibition menés in vitro sur l’enzyme isolée sont encourageants. Il en va autrement pour les tests menés sur le parasite Plasmodium falciparum. Lors de ceux-ci, des globules rouges colonisés par le parasite sont incubés en présence ou non de l’inhibiteur. Au bout de huit heures, les chercheurs comptent le nombre de globules rouges nouvellement infestés, afin de déterminer l’efficacité de l’inhibiteur.
Or à ce jour, l’IC50 (dose nécessaire à inhiber de moitié l’activité de SUB1 ou la croissance du parasite en culture) est d’environ 1.000 fois supérieure lors des tests menés sur les parasites à celle observée sur l’enzyme isolée. « Nous avons obtenu des composés d’intérêt, mais pas d’une efficacité suffisante. En termes d’affinité vis-à-vis de l’enzyme, nous avons atteint un niveau satisfaisant, mais les résultats ne suivent pas encore lorsque nous évaluons l’activité sur le parasite », constate Jean-François Hernandez. Une cause probable est une faible pénétration du composé à travers les membranes, ce qui limite son accès à sa cible. « Plus le composé est hydrophobe, mieux il traverse les membranes. Nous savons qu’il est possible de faire pénétrer des peptides de taille courte, mais à condition qu’ils soient de faible polarité ». De la même manière que les inhibiteurs de la protéase du virus de l’hépatite C, commercialisés depuis 2011, et forgés selon la même approche.
--------------------------------------------------------------------------------------------------------------
Ces recherches ont été financées en tout ou partie, par l’Agence Nationale de la Recherche (ANR) au titre de l'ANR SPIM - AAPG2019. Cette communication est réalisée et financée dans le cadre de l’appel à projet Sciences Avec et Pour la Société - Culture Scientifique Technique et Industrielle pour les projets JCJC et PRC des appels à projets génériques 2018-2019 (SAPS-CSTI JCJC et PRC AAPG 18/19).
- 1. La protéolyse limitée est une réaction consistant dans le clivage d’une protéine. Dans la cellule, c’est l’un des modes d’activation (ou d’inactivation) de nombreuses protéines. Cette réaction est catalysée par une protéase, qui peut elle-même être activée par protéolyse.
- 2. Une cascade enzymatique est une suite de réactions catalysées par des enzymes, s’activant de manière séquentielle. SUB1 induit ainsi une cascade de protéolyse, activant une série de protéases, aboutissant in fine à la destruction des membranes des cellules infectées.
- 3. M. Martinez, F. Batista, M. Maurel, A. Bouillon, L. Ortega, A.-M. Wehenkel, L. Le Chevalier-Sontag, A. Blondel, A. Haouz, J.-F. Hernandez, P. M. Alzari, J.-C. Barale, 3D structures of the Plasmodium vivax SUB1 subtilisin-like drug target reveal conformational changes to accommodate a substrate-derived alpha-ketoamide inhibitor, Acta Crystallogr. D Biol. Crystallogr. D79 (2023) 721-734. DOI: 10.1107/S2059798323004710
- 4. M. Martinez, F. Batista, M. Maurel, A. Bouillon, L. Ortega, A.-M. Wehenkel, L. Le Chevalier-Sontag, A. Blondel, A. Haouz, J.-F. Hernandez, P. M. Alzari, J.-C. Barale, 3D structures of the Plasmodium vivax SUB1 subtilisin-like drug target reveal conformational changes to accommodate a substrate-derived alpha-ketoamide inhibitor, Acta Crystallogr. D Biol. Crystallogr. D79 (2023) 721-734. DOI: 10.1107/S2059798323004710




Commentaires
Connectez-vous, rejoignez la communauté
du journal CNRS